

PhD, MCU-PH, MD, PhD, PU-PH, PharmD
slim.fourati@aphp.fr stephane.chevaliez@aphp.fr
Recherche clinique et translationnelle sur la résistance du VHC aux antiviraux à action directe (AADs)
L'arrivée sur le marché de nouveaux antiviraux actifs contre le virus de l'hépatite C (VHC) implique un fort investissement dans le développement de techniques innovantes permettant la recherche de substitutions amino acidiques associées à la résistance (resistance-associated substitutions, RASs) et la caractérisation phénotypique des conséquences de ces substitutions sur la sensibilité des souches virales correspondantes à l'antiviral. Les tests de résistance génotypique que nous avons mis en place et leur interprétation nous permettent de guider dans leurs décisions thérapeutiques les hépatologues, infectiologues et tous les cliniciens amenés à prendre en charge des patients atteints d'hépatite chronique C.
Nos travaux de recherche translationnelle concernent le retraitement, parfois complexe, des patients en échec de traitement par AADs exposés à un inhibiteur de la protéine non structurale NS5A. Ces échecs sont le plus souvent associés à la sélection de RASs localisés dans la séquence de la protéine NS5A, conférant le plus souvent une résistance croisée vis-à-vis de l'ensemble des inhibiteurs de la protéine NS5A disponibles (daclatasvir, ledipasvir, ombitasvir, elbasvir, pibrentasvir et velpatasvir). Ces RASs NS5A n'altèrent pas la capacité réplicative virale pour la majorité d'entre elles et ont la particularité de persister plusieurs années, peut-être pour toujours, après l'arrêt du traitement, rendant les approches de retraitement complexes.
Les principes généraux du retraitement consistent à utiliser une combinaison fondée sur le sofosbuvir (inhibiteur nucléotidique de la protéine NS5B dont la barrière contre la résistance est élevée) et une ou plusieurs autres molécules, idéalement sans résistance croisée avec celle(s) précédemment administrée(s) (Recommandations EASL pour le traitement de l'Hépatite C 2016, J Hepatol 2017). Notre centre est particulièrement impliqué dans l'évaluation de nouvelles stratégies de retraitement en cas de multi-échec de traitements par AADs. Nous avons par exemple récemment caractérisé les profils de résistance observés chez des patients en multi-échec thérapeutique recevant une triple combinaison de différentes classes d'AADs [sofosbuvir, daclatasvir, simeprevir (inhibiteur de protéase NS3) et ribavirine]. Les résultats ont montré la circulation de variants multi-résistants aux différentes classes d'antiviraux (conduisant parfois à une impasse thérapeutique).
La dynamique des variants résistants aux AADs est également étudiée dans le cadre d'études pilotes évaluant l'utilisation de nouvelles combinaisons thérapeutiques chez des malades en multi-échec thérapeutique comprenant:
Le deuxième sujet de nos activités de recherche porte sur l'optimisation des stratégies de traitement en termes de simplicité et de durée, afin d'améliorer l'adhésion du patient au traitement et éventuellement de diminuer les coûts. Dans ce cadre, nous avons récemment montré, à partir d'une cohorte de 221 patients cirrhotiques (cirrhose compensée sans épisode antérieur de décompensation) infectés par un VHC de génotype 1 ou 4 que les patients pour lesquels aucune RAS n'était détectable avant traitement (séquençage Sanger) pouvaient être traités pendant 12 semaines par une combinaison sans ribavirine associant le sofosbuvir et un inhibiteur de NS5A. En revanche, ce schéma thérapeutique n'était pas optimal chez les patients cirrhotiques avec RASs NS5A avant traitement, suggérant l'intérêt de la ribavirine chez ces patients.
Plateforme phénotypique d'étude de la résistance du VHC aux DAAs
La plateforme phénotypique d'étude de la résistance du VHC aux AADs a été mise en place en 2014, dans le cadre des activités du Centre National de Référence des Hépatites Virales B, C et Delta et de l'Observatoire National de la Résistance du VHC aux AADs adossé à la cohorte HEPATHER, financée par l'ANRS (ANRS CO22). Cette plate-forme permet d'identifier de nouveaux profils de résistance aux AADs afin de guider les futures stratégies thérapeutiques en cas d'échec virologique.
De nouvelles substitutions amino-acidiques sont introduites par mutagénèse dirigée dans différents réplicons subgénomiques (génotypes 1 à 5). Le réplicon VHC est fondé sur une construction chimérique bicistronique organisée de la manière suivante : un domaine comprenant l'extrémité 5' non codante du VHC, incluant l'IRES (site d'entrée interne du ribosome), le gène de résistance à la néomycine (G418), un gène rapporteur luciférase, puis un deuxième domaine comprenant l'IRES du virus EMCV (encephalomyocarditis virus) qui dirige la traduction des protéines non structurales du VHC ; la construction se termine avec l'extrémité 3' non codante du génome viral. Les réplicons subgénomiques sont dépourvus des séquences codant les protéines structurales p7 et NS2, qui ne sont pas nécessaires à leur réplication.
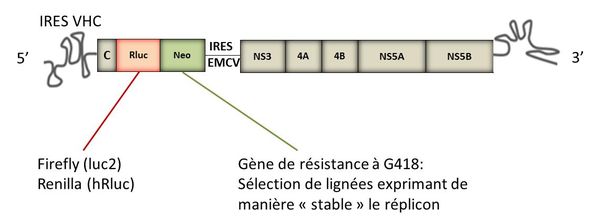
Structure d'un réplicon subgénomique du VHC
Les substitutions amino acidiques candidates sont introduites par mutagénèse dirigée sur un (ou plusieurs) plasmide(s) et l'analyse phénotypique est réalisée à partir de modèles transitoires (réplicons de sous-type 1a, 1b et 2a) ou de modèles dits " stables " (réplicons de génotypes 3, 4 ou 5) dans des lignées d'hépatomes humains, les cellules Huh-7.5.
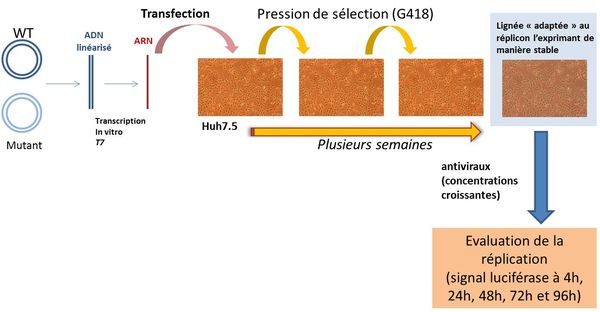
Mesure de la réplication d'un modèle de réplicon stable
Après mutagénèse dirigée et linéarisation du (ou des) plasmide(s), celui-ci (ceux-ci) est (sont) transcrit(s) in vitro via une ARN polymérase T7. L'ARN du réplicon subgénomique ainsi généré est transfecté dans les cellules Huh-7.5. Après stabilisation (pour les génotypes 3, 4 ou 5), les tests phénotypiques sont effectués en présence de concentrations croissantes d'antiviraux. L'inhibition de la réplication virale est évaluée par la mesure de l'activité luciférase en présence de différentes concentrations d'antiviraux, et ce à différents temps post-traitement. Le niveau de résistance des différents mutants est mesuré par l'étude de la concentration efficace 50% (CE50) et de la concentration efficace 90% (CE90), qui correspondent aux concentrations de l'antiviral permettant d'inhiber la réplication virale respectivement de 50% et de 90%.
Analyse de la dynamique d'évolution des quasi-espèces virales sous traitement et au moment de l'échec thérapeutique par analyse phylogénique de génomes complets à l'aide d'une approche de métagénomique shotgun
Notre équipe travaille sur la caractérisation de l'évolution des quasi-espèces virales par l'analyse longitudinale de séquences de génomes complets du VHC (majoritaires et minoritaires) avant, pendant et après traitement (en cas d'échec). Ce travail, financé par l'ANRS, a pour objectif de caractériser avec précision les dynamiques d'évolution des quasi-espèces virales au cours du traitement et après son arrêt (analyse de génome entier par approche " shotgun metagenomic "), et ainsi d'idendifier de nouvelles substitutions situées en dehors des régions ciblées par les AADs. Les nouvelles substitutions seront introduites par mutagénèse dirigée dans différents réplicons et testées vis-à-vis de différents antiviraux pour mettre en évidence le rôle éventuel " compensatoire " ou " résistant " conféré par ces substitutions, ainsi que lacapacité réplicative des réplicons correspondants.
Plateforme phénotypique d'étude de la sensibilité du VHB aux antiviraux
Une plateforme d'étude de la sensibilité phénotypique du VHB aux antiviraux a été récemment mise en place au laboratoire. Cette plateforme a pour objectifs : (i) de caractériser de nouvelles substitutions associées la résistance du VHB aux différents inhibiteurs de la transcriptase inverse et aux futures molécules en développement thérapeutique ; (ii) de cribler de nouvelles molécules capables d'inhiber la réplication virale et/ou d'inhiber d'autres étapes du cycle viral.
Le prototype utilisé est un plasmide codant le génome du VHB de génotype D de sous-type 1 sous le contrôle d'un promoteur CMV (Nassal et al, 1992).
Sofosbuvir-Daclatasvir-Simeprevir Plus Ribavirin in Direct-Acting Antiviral-Experienced Patients With Hepatitis C
Clin Infect Dis. 2017
Retreatment with sofosbuvir and simeprevir of patients with hepatitis C virus genotype 1 or 4 who previously failed a daclatasvir-containing regimen
Hepatology. 2016
Viral kinetics analysis and virological characterization of treatment failures in patients with chronic hepatitis C treated with sofosbuvir and an NS5A inhibitor
Alimentary Pharmacology Therapeutics 2017
Recent advances in understanding and diagnosing hepatitis B virus infection
F1000Res. 2016
Virologic Tools for HCV Drug Resistance Testing
Viruses. 2015