
Dr Christophe RODRIGUEZ, Responsable
"La métagénomique, au cœur du dépistage et de la caractérisation des pathogènes microbiens."
La plateforme de séquençage à haut débit (next-generation sequencing, NGS) a été créée en 2012 à l'initiative de l'Institut de Recherche Biomédicale (IMRB), centre de recherche INSERM de la faculté de médecine de Créteil, et du Pôle de Biologie et de Pathologie du Groupe Hospitalier Henri Mondor (Assistance Publique-Hôpitaux de Paris), soutenue par un financement INTER-DIM (Région Ile-de-France) et des financements spécifiques de l'Université Paris-Est Créteil, de l'Assistance Publique-Hôpitaux de Paris et de l'Agence Nationale de Recherche sur le SIDA et les Hépatites Virales (ANRS). Positionnée dès l'origine autour d'un axe microbiologique médical et de recherche, la plateforme occupe aujourd'hui une place importante dans le développement des techniques de NGS pour le diagnostic et la caractérisation des pathogènes microbiens.
L'émergence constante de nouvelles infections, favorisées par la globalisation, les voyages, le réchauffement climatique, posent la question du diagnostic des infections devant des symptômes peu spécifiques, dans des contextes peu évocateurs, sans pré-connaissance du germe recherché. Le développement de tests diagnostiques spécifiques des infections recherchées est par ailleurs coûteux, long à mettre en œuvre et souvent disponible trop tardivement. Le laboratoire s'est spécialisé dans le développement et l'application d'un test diagnostique d'infection " universel ", fondé sur une approche métagénomique. La métagénomique consiste à établir le répertoire de l'ensemble des espèces en présence dans un échantillon donné. Une première application a été la métagénomique ciblée (16S ou ITS) pour les bactéries ou les champignons, qui ne permettait cependant pas la recherche de virus à ADN ou ARN.
Notre laboratoire a développé un process complet de " shotgun metagenomic " permettant :
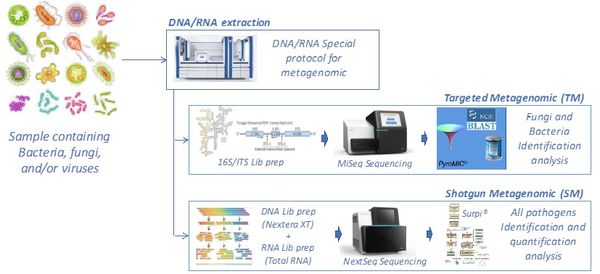
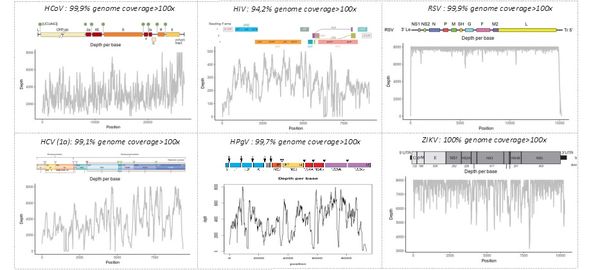
La technique " shotgun metagenomic " a été validée sur des échantillons mono- ou polymicrobiens contenant des pathogènes bactériens, fongiques et/ou viraux. La figure ci-dessous montre la capacité à identifier les séquences de génomes entiers de différents virus provenant d'échantillons humains, incluant le VIH, le virus de l'hépatite C (VHC), le pegivirus humain de type 1 (HPgV), le virus zika (ZIKV), ou le virus respiratoire syncitial (VRS), ou de culture cellulaire (coronavirus humain 229E, HCoV). Pour chaque région du génome viral, la profonduer indique le nombre de séquences générées, permettant l'identification du pathogène par comparaison avec les banques de séquences.
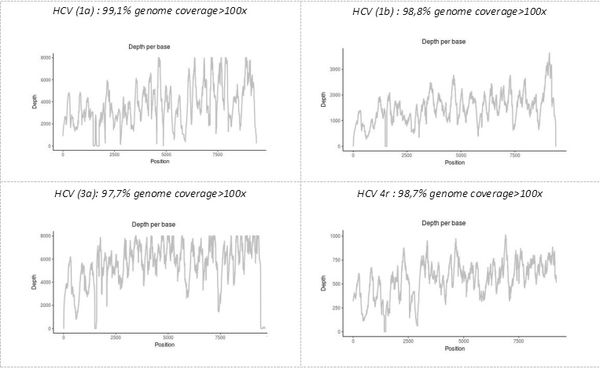
L'identification précise du ou des pathogènes ainsi que la mise en évidence de certaines de leurs caractéristiques génomiques ayant une incidence fonctionnelle est un autre atout de la technique de " shotgun metagenomic " développée sur la plateforme. Le séquençage en une seule expérimentation du génome complet de chaque pathogène présent permet d'obtenir l'ensemble des données génétiques, éventuellement à un niveau de variant minoritaires. Par exemple, la technique permet le génotypage et le sous-typage, ou la détection de variants viraux résistants dans des échantillons d'intérêts provenant du CNR des Hépatites Virales B, C et Delta.
Dans le cadre de ses missions de veille technologique, la plateforme teste également régulièrement de nouveaux équipements et/ou kits de NGS, en particulier dans le domaine du diagnostic et du typage.
Les techniques de NGS permettent, dans le cadre de projets de recherche, de caractériser les modifications observées chez l'hôte dans différentes situations d'infection, avec ou sans traitement anti-infectieux. Les méthodes développées comprennent le RNA-Seq, le micro-RNA Sequencing et la transcriptomique. En lien avec le groupe travaillant sur le développement de nouvelles approches antivirales à large spectre, ces outils apportent de précieux renseignements pour l'identification des cibles antivirales et la compréhension des mécanismes d'action des antiviraux.